Forschung
Forschungsschwerpunkte
Tetrapyrrole und ihre Biosynthese
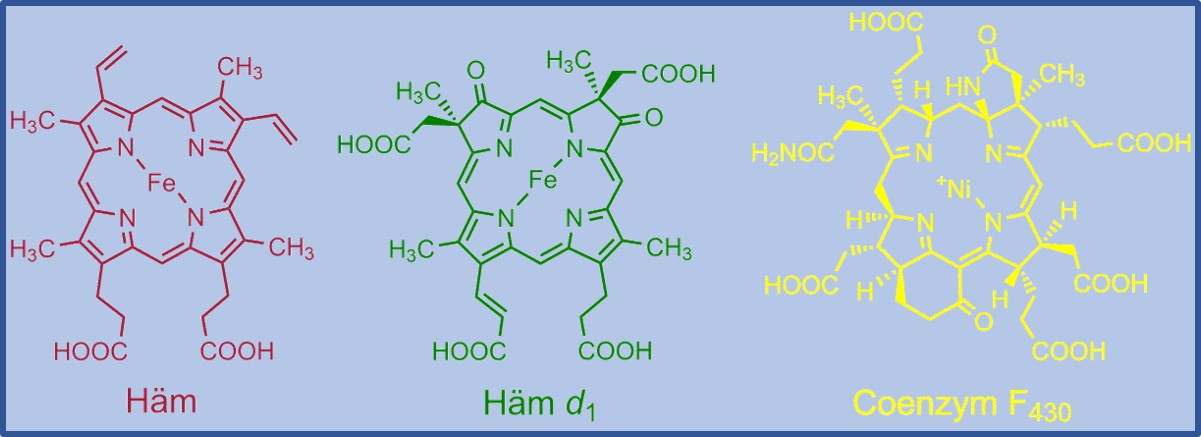
Tetrapyrrole, wie z.B. Häm, Chlorophyll oder Cobalamin, sind in der Natur ubiquitär vorkommende Moleküle mit einem breiten Spektrum an essentiellen Funktionen für nahezu alle Organismen auf der Erde. Weitere Beispiele sind das Häm d1, das nur in manchen denitrifizierenden Bakterien vorkommt, und Coenzym F430, das in allen methanbildenden Archaeen essentiell ist. Die Abbildung 1 zeigt die Strukturen dieser Moleküle.
Enzyme der Sirohäm-abhängigen Häm- und Häm d1 Biosynthese
Sowohl die Hämbiosynthese in Archaeen und sulfatreduzierenden Bakterien als auch die Häm d1 Biosynthese in manchen denitrifizierenden Bakterien verlaufen über die gemeinsamen Intermediate Sirohäm und 12,18-Didecarboxysirohäm. Anschließend unterscheiden sich die weiteren Reaktionsschritte und beteiligten Enzyme zwischen den beiden Wegen, wie es die Abbildung 2 zeigt. Derzeit untersuchen wir insbesondere die Hämsynthase AhbD, die den letzten Schritt der Hämbiosynthese katalysiert, sowie NirJ aus der Häm d1 Biosynthese. Bei beiden Enzymen handelt es sich um Radical SAM Enzyme, deren radikalische Katalysemechanismen wir aufklären möchten.
mehr unter: Layer (2021) BBA - Molecular Cell Research, 1868, 118861
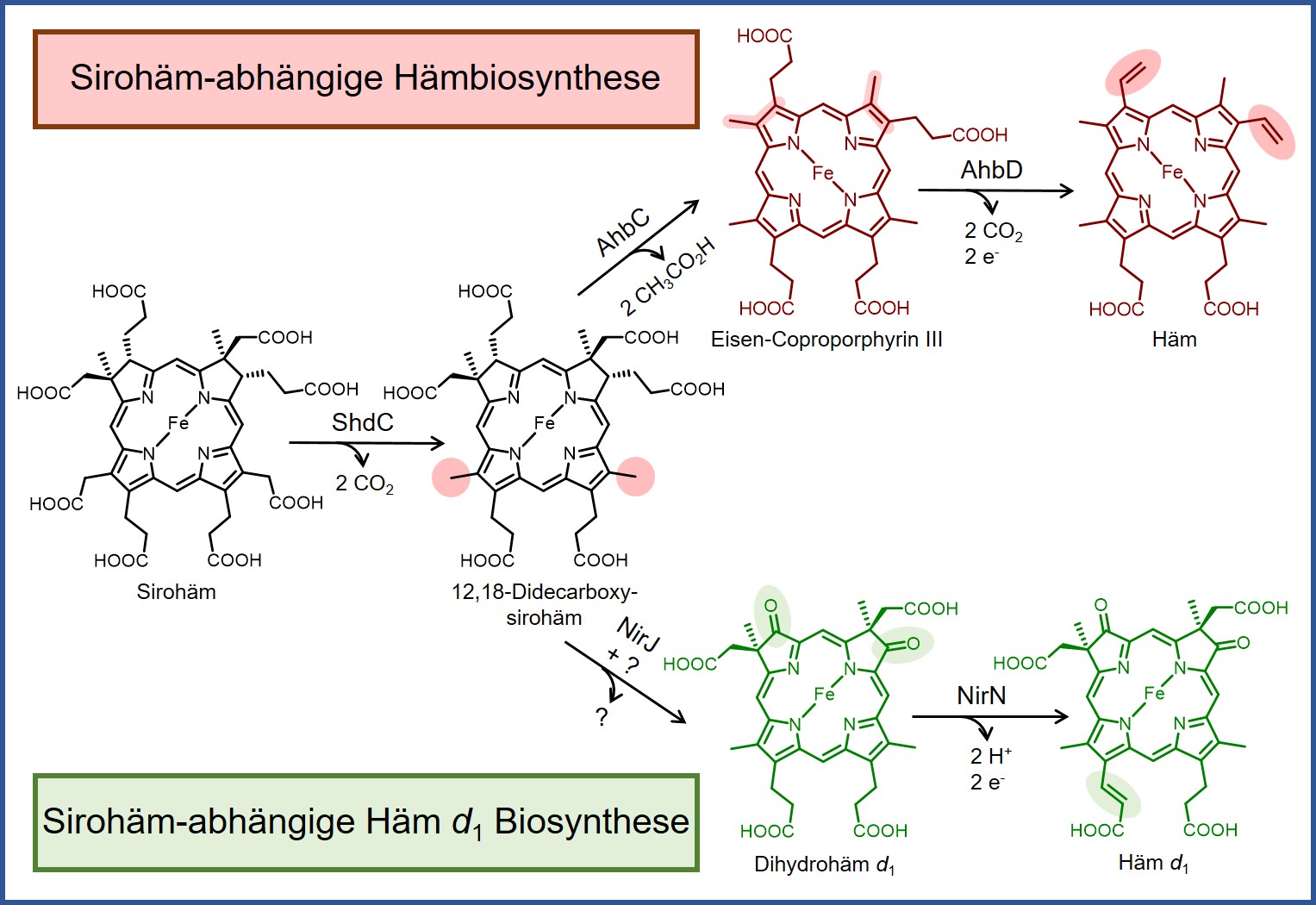
Abbildung 2: Biosynthese von Häm in Archaeen und sulfatreduzierenden Bakterien sowie Häm d1 Biosynthese in denitrifizierenden Bakterien.
Enzyme der Coenzym F430 Biosynthese
Das nickelhaltige Coenzym F430 ist der essentielle Cofaktor der Methyl-Coenzym M Reduktase methanbildender sowie anaerob methanoxidierender Archaeen und damit sowohl an der Bildung als auch am Verbrauch von Methan in der Natur wesentlich beteiligt. Die Biosynthese des Coenzyms F430 ist in Abbildung 3 gezeigt. Derzeit untersuchen wir die Reduktase CfbC/D, die verwandt zur Nitrogenase ist und eine bemerkenswerte 6-Elektronen-Reduktion des Tetrapyrrolringsystems unter Einführung sieben neuer Stereozentren katalysiert.
mehr unter: Moore et al. (2017) Nature, 543, 78-82
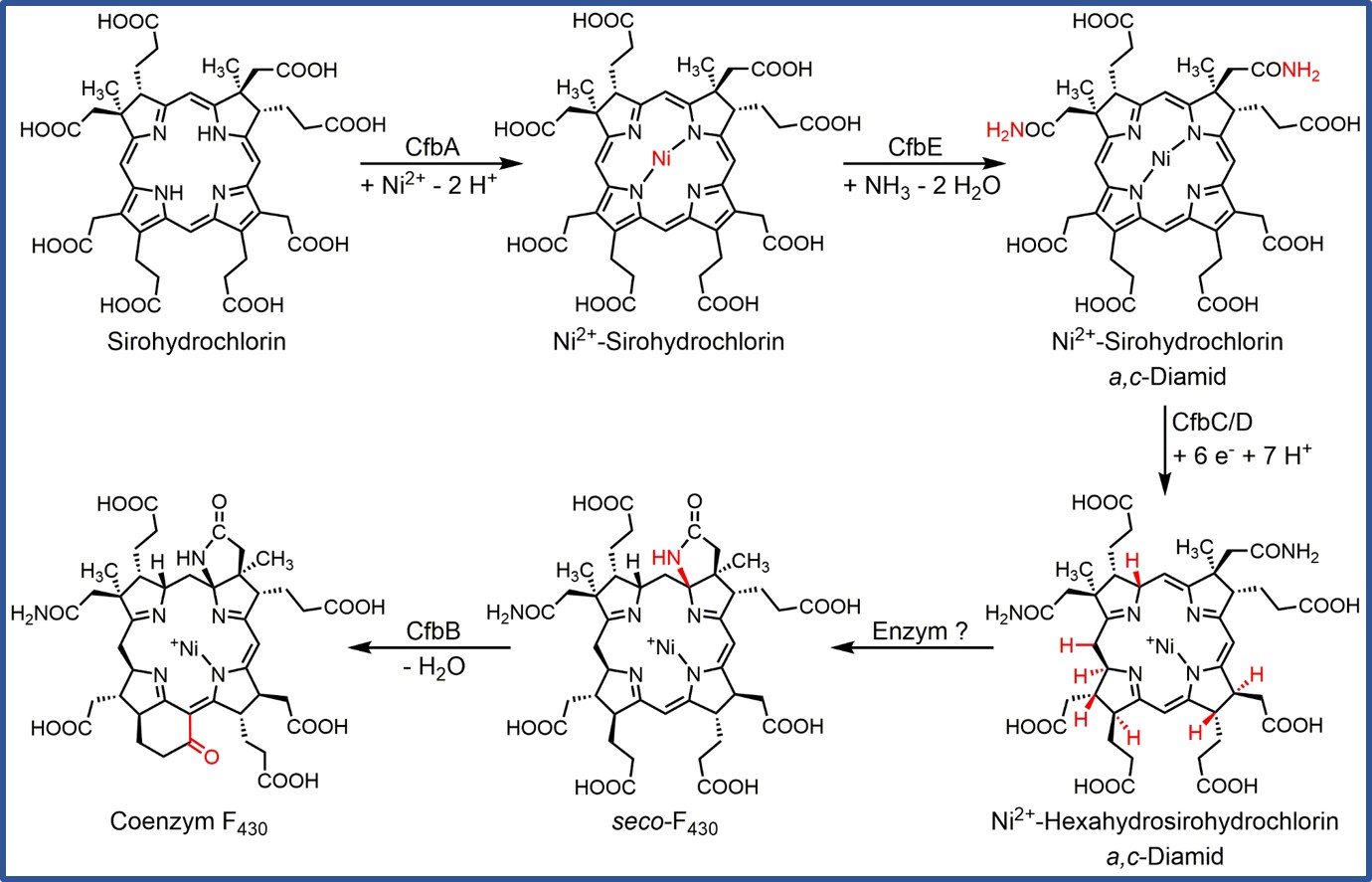
Abbildung 3: Biosynthese von Coenzym F430.
Posttranslationale Modifikationen der Methyl-Coenzym M Reduktase
Die Methyl-Coenzym M Reduktase methanbildender sowie anaerob methanoxidierender Archaeen trägt einige ungewöhnlich modifizierte Aminosäuren, darunter die einzigartigen C-methylierten Aminosäuren 5-C-(S)-Methylarginin und 2-C-(S)-Methylglutamin (Abbildung 4). Diese Modifizierungen werden von zwei cobalaminabhängigen Radical SAM Methyltransferasen eingeführt, welche wir in unserer Gruppe untersuchen.
mehr unter: Gagsteiger et al. (2022) Angew. Chem. Int. Ed., 61, e202204198
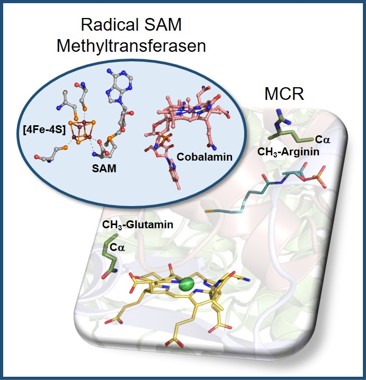
Abbildung 4: Cobalaminabhängige Radical SAM Methyltransferasen katalysieren die Einführung der Methylarginin- und Methylglutaminmodifikationen in die Methyl-Coenzym M Reduktase (MCR).

