Forschung
Forschungsschwerpunkte unserer Arbeitsgruppe
Das wichtigste wissenschaftliche Ziel unserer Arbeitsgruppe ist es, neue antibiotisch wirksame Verbindungen zu generieren oder in der Natur zu finden und Verfahren zu entwickeln diese Naturstoffe in Mengen bis zu 100 mg zu produzieren. Um diese Zeil zu erreichen bearbeiten wir folgende Projekte:
Suche nach neuen Aktinomyceten in Insekten
Das Mikrobiom von Insekten ist noch nicht besonders intensiv erforscht. Trotzdem ist bekannt, dass Insekten von z.T. recht spezifischen Bakterien besiedelt sind. Kürzlich haben wir Projekte begonnen Aktinomyceten in Insekten zu identifizieren und zu charakterisieren. Im Mittelpunkt stehen Bienen und Borkenkäfer, die wir zusammen mit einem Freiburger Imker bzw. einer Freiburger Försterin untersuchen.
Aufwecken „stiller Biosynthesegencluster“ unter Einsatz von regulatorischen Genen
Antibiotika-Biosynthese-Gene in Aktinomyceten liegen alle dicht zusammen auf einem kleinen Abschnitt des Chromosoms vor. Man spricht von einem "Biosynthese-Gencluster". Von etwa 30 Genclustern, die in einem Genom eines Aktinomyceten lokalisiert sind, werden üblicherweise nur eine kleine Anzahl zur Expression gebracht. Um die nicht exprimierten (stillen) Gencluster zu aktivieren untersuchen wir die Funktion regulatorischer Gene, um diese dann gezielt zur Aktivierung der Cluster einzusetzen. Im Mittelpunkt unserer Untersuchungen stehen derzeit c-di-GMP bildende (Diguanylat Cylasen) und abbauende (Phosphodiesterasen) Enzyme (Abbildung 1).
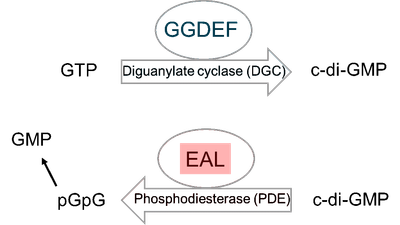
Abbildung 1: c-di-GMP bildende und abbauende Enzyme
Charakterisierung ungewöhnlicher Biosyntheseenzyme
Immer wieder findet man in Biosynthesegenclustern Enzyme, die ungewöhnliche Reaktionen katalysieren. Ein Beispiel ist das Enzym RslO9, das eine Umlagerung während der Rishirilid-Biosnthese katalysiert (Abbildung 2). Die Aufklärung der genauen Funktion dieser Enzyme ist die Grundlage dafür, dass derartige Enzyme später als Werkzeuge in der Biotechnologie verwenden werden können.
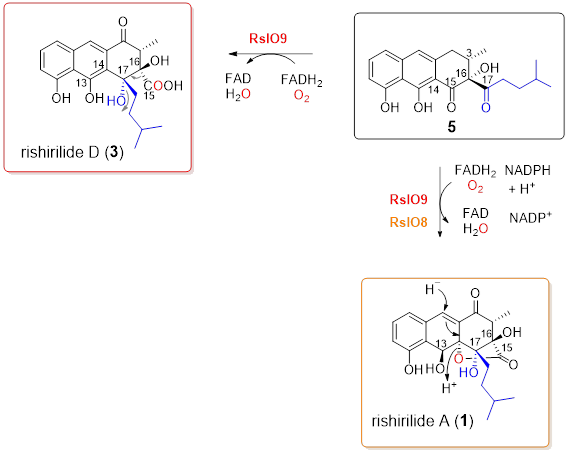
Abbildung 2: Baeyer-Villiger-Oxidation katalysiert durch RslO9
Herstellung neuer Arzneistoffe mittels „Kombinatorischer Biosynthese"
Biosynthesergene können gezielt eingesetzt werden, um neue Antibiotika herzustellen. Viele Jahre lang haben wir Glycosyltransferase-Gene verwendet um neue glycosidierte Arzneistoffe zu erzeugen. Dabei haben wir auch selbst designte Glycosyltransferasegene eingesetzt, deren Substratspezifität verändert war. Derzeit fokussieren wir uns auf Oxygenasegene, deren Potential für die „Kombinatorische Biosynthese“ aber derzeit noch erforscht wird.
Herstellung eines Produzenten, der 24-Desmethylrifamycin in großen Mengen herstellen kann
Das Antibiotikum Rifamycin kann zur Behandlung von Tuberkulose eingesetzt werden. Leider sind in vielen Ländern der Welt die Erreger (Mycobakterium tuberculosis) der Tuberkulose resistent gegen das Antibiotikum. Prof. Dr. R. Lal aus New Delhi entwickelte einen Stamm, der 24-Desmethylrifamycin (Abbildung 3) produziert. Diese Verbindung kann partialsynthetisch zu Derivaten umgesetzt werden, die auch gegen resistente Keime aktiv sind. Leider wird das Antibiotikum nur in sehr geringen Mengen hergestellt. Derzeit versuchen wir den Stamm so zu modifizieren, dass er in der Lage ist 24-Desmethylrifamycin in großen Mengen herzustellen.
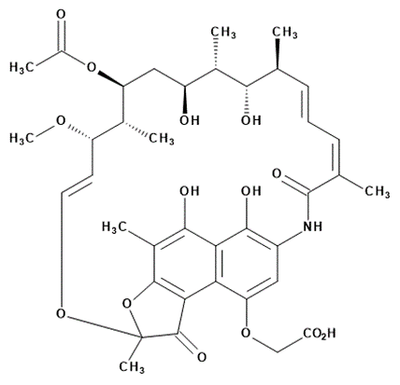
Abbildung 3: Struktur des Antibiotikums 24-Desmethylrifamycin B

